La sintesi del DNA avviene durante la fase S del ciclo cellulare attraverso la duplicazione del DNA parentale e la formazione di due molecole di DNA identiche distribuite poi nelle due cellule figlie in seguito alla divisione cellulare.
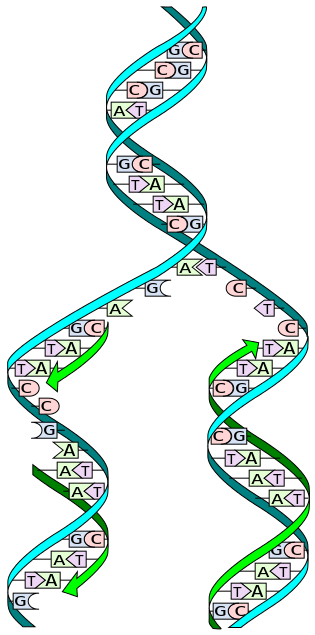
In pratica, ciascuno dei due filamenti serve da stampo per la sintesi di un nuovo filamento complementare, per cui la cellula figlia avrà un filamento originale derivante dal DNA parentale, e un filamento complementare neosintetizzato, per questo motivo la replicazione del DNA è definita semi-conservativa.
La sintesi del DNA viene controllata da vari enzimi e proteine, tra cui la DNA polimerasi, diversa per ogni organismo.
Nelle cellule procariotiche esistono tre tipi di DNA polimerasi: la DNA polimerasi III che è deputata alla replicazione del DNA con attività di sintesi in direzione 5’ -> 3’ con velocità di circa 1000 nucleotidi al secondo; la DNA polimerasi I ha il compito di replicazione e riparazione del filamento di DNA e la DNA polimerasi II che ha solo il compito di riparare il filamento. La riparazione è dovuta ad attività di esonucleasi 3’ -> 5’ con correzione degli errori che avvengono durante la sintesi.
Nelle cellule eucariotiche esistono invece tre principali classi di DNA polimerasi:
- Le DNA polimerasi DNA-dipendenti ad alta fedeltà: definite “DNA-dipendenti” perché usano il filamento stampo di DNA per la sintesi del filamento complementare, e definite “ad alta fedeltà” perché sono dotate di attività esonucleasica 3’ -> 5’ di correzione degli errori con alta fedeltà di replicazione (1 errore ogni 10 nucleotidi copiati). Nel nucleo queste polimerasi sono la DNA polimerasi α e δ con velocità di sintesi pari a circa 50 nucleotidi al secondo, mentre la riparazione è dovuta alla DNA polimerasi ε che è in grado di identificare le basi disposte in maniera errata, tagliare il segmento di DNA con l’errore e sintetizzare il DNA corretto. Nei mitocondri, invece, la sintesi e riparazione del DNA sono modulate dalla DNA polimerasi γ.
- Le DNA polimerasi DNA-dipendenti a bassa fedeltà: sono DNA polimerasi che hanno un tasso di errore 20.000 volte maggiore alle DNA polimerasi ad alta fedeltà.
- Le DNA polimerasi RNA-dipendenti (trascrittasi inverse): sono enzimi che sintetizzano il DNA a partire da uno stampo di RNA, come l’enzima telomerasi, deputato alla sintesi dei telomeri.
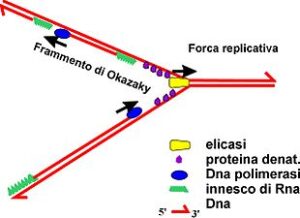
Nelle fasi iniziali della duplicazione, si ha l’intervento dell’enzima topoisomerasi II, che taglia un filamento del DNA favorendone lo svolgimento.
La replicazione avviene poi in punti specifici detti origine di replicazione. Nei procarioti si ha solo un’origine, mentre nelle cellule eucariotiche sono attive più origini di replicazione contemporaneamente, motivo per cui la fase S ha una breve durata (1-2 h).
A livello delle origini viene a formarsi la forcella di replicazione, un punto di biforcazione della doppia elica parentale, in cui interviene l’enzima elicasi che determina il progressivo srotolamento della doppia elica del DNA parentale, favorendo la separazione dei due filamenti di DNA con formazione del filamento guida (leading strand) e del filamento lento (lagging strand), così chiamato perché la sua sintesi avviene in modo più lento rispetto a quella del filamento guida.
Nella sintesi del DNA intervengono anche le proteine SSB (Single strand-binding protein) che si legano ai filamenti denaturati impedendo che questi possano riavvolgersi a formare la doppia elica che bloccherebbe la replicazione.
La sintesi dei filamenti si deve all’intervento dell’enzima DNA polimerasi α, che usa i precursori dNTP, cioè i deossinucleosidi trifosfato (dATP, dCTP, dGTP, dTTP), da cui derivano i nucleotidi. Tuttavia, prima di iniziare la sintesi, è necessario l’intervento dell’enzima primasi che catalizza la sintesi di un primer innesco, un corto filamento di RNA costituito da 3-5 nucleotidi che presenta un gruppo –OH libero all’estremità 3’ dove la DNA polimerasi aggiunge nuovi nucleotidi complementari al filamento stampo, unendo i nucleotidi in successione mediante legami fosfodiesterici in direzione 5’->3’. L’energia necessaria alla reazione è fornita dall’idrolisi del pirofosfato (PPi) ad opera di una pirofosfatasi.
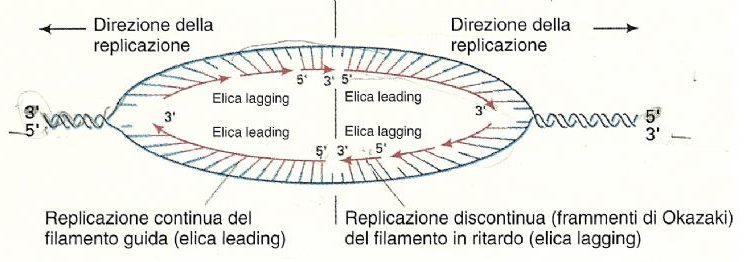
La sintesi del DNA è asimmetrica e semi-discontinua perché il DNA è costituito da 2 filamenti complementari e antiparalleli, il filamento lagging e il filamento leading.
La sintesi del filamento guida avviene in direzione 5’->3’, nella direzione di movimento della forcella di replicazione, con sintesi continua perché la DNA polimerasi aggiunge nucleotidi in successione al gruppo –OH libero in 3’.
La sintesi del filamento lento avviene sempre in direzione 5’->3’, ma in direzione opposta al movimento della forcella di replicazione e in modo discontinuo perché viene sintetizzato in piccoli segmenti detti frammenti di Okazaki, lunghi 100-1000 nucleotidi, ciascuno sintetizzato a partire da un primer a RNA. Questi frammenti vengono poi uniti dall’enzima DNA ligasi che catalizza la formazione di un legame fosfodiestere tra il gruppo –OH 3’ dell’estremità di un frammento di Okazaki e il gruppo fosfato 5’ del frammento di Okazaki adiacente, formando un filamento continuo.
Fonte: Wikipedia



