L’isoelettrofocalizzazione o isoelettrofocusing (IEF) è una tecnica che permette una separazione delle molecole proteiche o di peptidi, che sono più piccoli delle proteine, in base al punto isoelettrico. La separazione delle varie molecole proteiche, caratterizzate ognuna del proprio punto isoelettrico, dipende essenzialmente dalla composizione amminoacidica che ci permette di distinguere le proteine acide, basiche e p anche neutre che comunque presentano un valore isoelettrico compreso tra 6 e 7.5/8.
Per ottenere la separazione bisogna caricare il campione su uno STRIP, una colonnina oppure una striscia di gel di poliacrilammide, che è stata premiscelata con una soluzione di anfoliti, sostanze ognuna caratterizzata da una stessa carica che si dispongono lungo la strip di gel a formare un gradiente di pH, Queste strip commercialmente sono presente in vari tipi: intervallo di pH basico, intervallo di pH acido, a seconda della proteina che dobbiamo andare a separare. Quindi si applica con la pipetta il campione e si sottopone lo strip di gel alla migrazione applicando un campo elettrico (in quanto si tratta sempre di una tecnica elettroforetica che utilizza la migrazione di una particella carica sottoposta ad un campo elettrico).
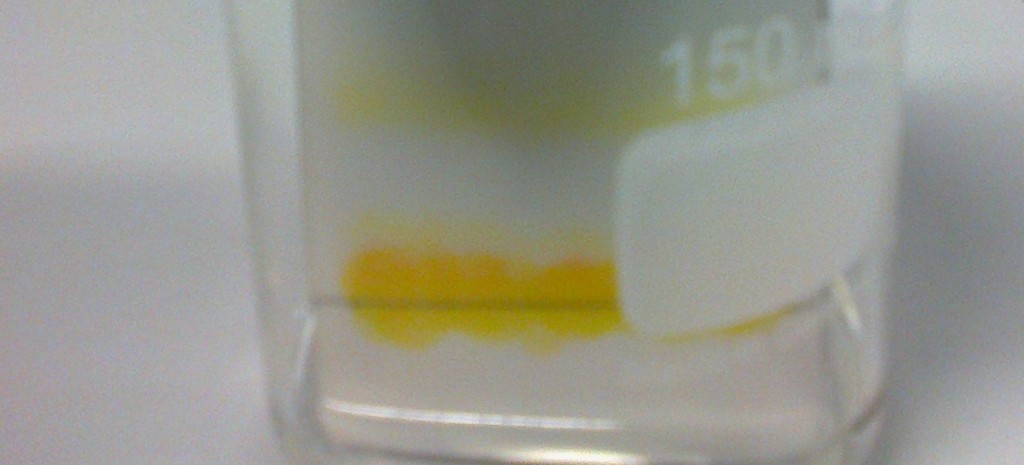
La differenza, rispetto alle altre tecniche, soprattutto la SDS-PAGE, è che con essa otteniamo la separazione delle molecole proteiche, dato che sono tutte cariche negativamente, in base al peso molecolare, alla massa molecolare della macromolecola. In questo caso, invece, con la miscela di proteine che abbiamo depositato sull’inizio della strip, in uno dei due punti, le particelle migreranno fino a quando non giungeranno in quella regione del gel il cui pH corrisponde al punto isoelettrico della proteina. Quindi possiamo vedere una separazione di bande di questo tipo. Questo si ottiene after staining, cioè dopo la colorazione, in quanto noi non riusciamo a seguire la corsa.
Elettroforesi bidimensionale
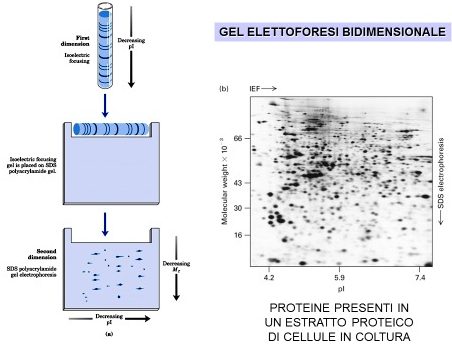
Un campione di proteine può essere stratificato su un’estremità di una striscia di gel, dove è presente un gradiente stabile di pH. In alternativa, lo stesso campione di proteine, sospeso in una soluzione di anfoliti, può essere, invece, utilizzato per reidratare una striscia di gel disidratata. Viene applicato il campo elettrico, dopo la colorazione le proteine risultano distribuite lungo il gradiente di pH secondo il loro valore di punto isoelettrico. Questa separazione in isoelettrofocalizzazione, può essere seguita da una seconda tappa elettroforetica che costituisce l’esempio di elettroforesi bidimensionale.
Dopo opportuni passaggi di solubilizzazione delle proteine che si trovano all’interno della matrice porosa di poliacrilammide, questa striscia di gel montata sul secondo gel viene sottoposta ad un campo elettrico e questo serve per ottenere la separazione delle proteine che si trovano in ogni banda proteica ottenuta con la prima separazione. Tutto questo presuppone che ogni banda proteica che abbiano ottenuto con la prima separazione, è costituita da un certo numero di subunità. Con la seconda dimensione, otteniamo la separazione delle varie subunità e l’identificazione di tutte o quasi tutte le proteine che si trovano in ogni banda e che vengono quindi visualizzate poi nel gel di seconda dimensione, sempre dopo colorazione, come degli spot (ossia dei puntini, come infatti si può notare in questa parte a destra, ogni spot corrisponde ad una proteina).
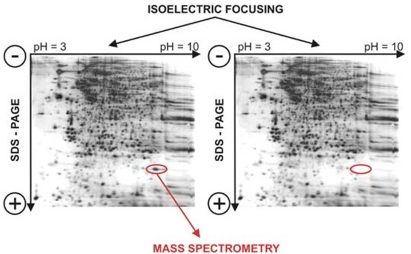
Paragonando il risultato della bidimensionale ottenuto dall’immagine di sinistra rispetto a quella di destra, ad occhio il ricercatore può notare che c’è la scomparsa di uno degli spot nel gel di destra; questo può corrispondere ad una regolazione di una proteina in uno stato patologico o in seguito ad un trattamento con un farmaco, dipende da quello che si sta studiando. Serve quindi come applicazione di uno screening iniziale per poter identificare la proteina coinvolta in quel determinato pathway o in una determinata via metabolica.
Per poter poi identificare la proteina specifica bisogna analizzare in dettaglio, come per esempio si può fare con la spettrometria di massa, che permette l’identificazione in base alla massa di una proteina e quindi di essere sicuri di quale proteina sia coinvolta nello studio che stiamo facendo. Spot che si trovano più in basso sono quelle che hanno un peso molecolare più basso a differenza di quelle che si trovano più in alto (stesso principio di separazione di altre tecniche).
Blue native PAGE
Un tipo molto particolare di elettroforesi bidimensionale è rappresentato dalla blue native PAGE (dove PAGE significa elettroforesi su gel poliacrilammide), ed è un tipo di tecnica elettroforetica che viene molto utilizzata dai mitocondriologi.
È sempre una tecnica elettroforetica bidimensionale, quindi c’è una prima dimensione che permette la separazione, nel caso dei mitocondri, dei complessi respiratori, e poi nella seconda dimensione ogni complesso viene suddiviso nelle varie subunità proteiche. Si chiama “blue” perché la sostanza fondamentale che viene utilizzata è il “blue comassie” (comassie blue G250) che colora di blu tutti campioni, anche i buffer di corsa che vengono utilizzati. Si chiama, invece, “native” perché è un’elettroforesi di tipo nativo, in quanto i complessi della catena respiratoria che vengono separati con questa tecnica devono necessariamente mantenere la propria conformazione nativa, infatti al termine della separazione dei vari complessi, si può anche effettuare un saggio di funzionalità. Per esempio, al complesso primo, che è il primo nel gel perché è il più pesante, se si aggiunge, in opportune condizioni sperimentali, il NADH che è il trasportatore di elettroni e protoni nel complesso primo, si osserva l’ossidazione del NADH a NAD+, a testimoniare la funzionalità dell’enzima, e così dicasi per gli altri complessi.
La conformazione nativa si può preservare trattando le nostre cellule, che derivano da una coltura cellulare o da un omogenato di tessuto, con degli agenti solubilizzanti che riescano a solubilizzare le membrane (però non troppo perché se fossero degli agenti troppo aggressivi porterebbero anche alla dissociazione dei complessi come quelli della catena respiratoria formati da un insieme di subunità). Quindi, per fare questo, utilizziamo dei detergenti blandi, che sciolgono le membrane, però preservano la struttura nativa, come la digitonina (un detergente blando che funziona formando dei pori sulle membrane biologiche, e può essere più o meno blando a seconda della concentrazione che noi utilizziamo). La digitonina svolge anche il ruolo di purificazione dei mitoplasti (un derivato dei mitocondri, un mitocondrio privato della membrana esterna costituito dalla membrana interna con i complessi della catena respiratoria che rimangono in conformazione nativa ancora sulla membrana).
La funzione delle altre sostanze come il comassie blu G250 e l’acido aminocaproico, è quella di conferire una leggera carica negativa ai complessi. L’acido aminocaproico è una sostanza acida che viene utilizzata sia nella fase di solubilizzazione che nella costituzione del gel. Anche il gel che viene utilizzato per mettere in pratica questo tipo di tecnica, è sempre un gel di poliacrilammide, però è un gel nativo. E’ assolutamente bandito l’SDS (sodio dodecil solfato) perché è l’agente denaturante più forte. Se i mitocondri vengono a contatto con l’SDS si denatura tutto e non riusciamo a venire a contatto con la conformazione nativa.
Questo tipo di elettroforesi viene utilizzata per i mitocondri ma anche per altri organuli cellulari, come i cloroplasti oppure complessi multienzimatici, come quelli della catena respiratoria. La distribuzione dei complessi è in base al peso molecolare. Il complesso I è quello che ha un peso molecolare maggiore, con 45 o più subunità, e poi a scalare complesso quinto, terzo, quarto e secondo.
Se vogliamo dissociare i vari complessi della catena nelle subunità mettiamo un medium di solubilizzazione che contiene agenti denaturanti tipo il β-SH, il β-mecarptoetanolo e si ottiene alla fine, dopo che il gel viene colorato, un pattern elettroforetico, in cui ogni singola banda corrisponde ad ogni singola subunità dei complessi. Non vengono visualizzate tutte e 45 le subunità del complesso primo, ma soltanto quelle che si trovano in questo intervallo di peso molecolare.
Un avanzamento nella letteratura scientifica ha dimostrato che i complessi della catena respiratoria sono organizzati in maniera associata, si trovano associati tra di loro in maniera tale che il passaggio degli elettroni da un complesso all’altro attraverso i trasportatori ausiliari, che sono il coenzima Q, o il citocromo C che collega il III col IV, sia ottimizzato per evitare perdite di energia, una situazione che comporterebbe l’aumento di produzione dei radicali liberi. Nonostante sia molto difficile preservare la conformazione di questi super complessi, è stato evidenziato che molte patologie umane sono collegate alla diminuzione dei super complessi. Tanto maggiore è la presenza dei super complessi, tanto più efficiente è la produzione energetica della cellula e ci sono alcune patologie in cui si assiste ad una progressiva degenerazione della cellula e quindi dell’organismo, in funzione della degenerazione mitocondriale misurata come diminuzione della costituzione dei super complessi. Essendo più attivi i super complessi, rispetto ai complessi separati, in una patologia mitocondriale diminuiscono e aumenta lo stato patologico.